Разработчик
ФГБНУ ФИЦ ИЦиГ СО РАН
Авторы
Левицкий В.Г., Цуканов А.В.
Краткая характеристика
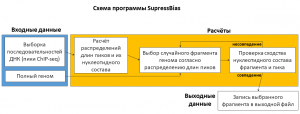
Программа SuppressBias предназначена для генерации выборки негативных последовательностей ДНК с целью подавления влияния фоновых мотивов обогащенных в геномной ДНК в целом при анализе обогащения мотивов в полногеномных данных массового секвенирования (например, результатах экспериментов ChIP-seq или RNA-seq). Для работы программе требуется позитивная выборка последовательностей ДНК, представляющих результат полногеномного эксперимента, и полный геном. Программа производит поиск в полном геноме последовательностей ДНК, соответствующих последовательностям позитивной выборки по длине и составу k-меров (коротких подпоследовательностей длины k). Программа содержит блок оценки распределения частот k-меров в позитивной выборке и блок генерации выборки негативных последовательностей ДНК. Программа генерирует выборку негативных последовательностей ДНК, в которой распределения длин и частот k-меров отражает таковые для позитивной выборки. Программа может учитывать k-меры длин от 1 до 4. Программа может быть использована для выявления обогащения отдельных мотивов или для поиска обогащённых мотивов с помощью подхода de novo.
Области возможного использования
Программа может быть использована в биоинформатике и генетике для выявления обогащения отдельных мотивов или для поиска обогащённых мотивов с помощью подхода de novo.
Степень готовности разработки к практическому применению
Программа готова к практическому применению.
Возможный технический и (или) экономический эффект
Экономия средств при анализе результатов полногеномных экспериментов.
Сравнительные характеристики с известными разработками
В РФ аналогов нет, за пределами РФ есть аналог https://doi.org/10.1093/bioinformatics/btaa928, однако его функциональные возможности ограничены.
Защита разработки
Свидетельство о регистрации №2022612715, зарегистрирована в Реестре ПрЭВМ 28.02.2022.