Разработчик
ФГБНУ ФИЦ ИЦиГ СО РАН
Авторы
Бобровских А. В., Зубаирова У. С., Дорошков А. В.
Краткая характеристика
База данных AraLightDEGs представляет собой платформу для хранения и анализа информации о дифференциально экспрессирующихся генах (ДЭГ) модельного растения Arabidopsis thaliana в ответ на избыточную освещенность. Данные были получены в результате систематического поиска, отбора и биоинформатической обработки 21 транскриптомного эксперимента, опубликованных в репозиториях NCBI (GEO, SRA, BioProject). Все данные прошли строгую проверку на соответствие критериям отбора, включая генотип Columbia (Col-0), фотосинтетические ткани и условия освещенности (контроль: 50–150 мкмольм⁻²с⁻¹; стресс: ≥ 500 мкмоль м⁻²с⁻¹) и контроль качества.
Обработка данных включала выравнивание прочтений на референсный геном, контроль качества, подсчет экспрессии и идентификацию ДЭГ с использованием современных биоинформатических инструментов (HISAT2, RNA-SeQC, featureCounts, edgeR). В результате было идентифицировано 218 335 ДЭГ, соответствующих 19 723 уникальным генам.
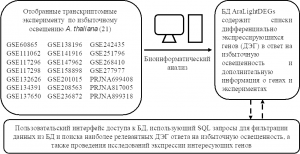
БД реализована с использованием СУБД MariaDB, её структура представлена на Рисунке 1. Пользовательский интерфейс БД, реализованный с использованием современных технологий (HTML5, CSS3, JavaScript, Flask), предоставляет удобный доступ к данным через веб-ресурсы ИЦиГ СО РАН (https://www.sysbio.ru/aralightdegs/). Разработанная БД позволяет регулярно обновлять её содержимое по мере поступления новых данных из публичных репозиториев (таких как NCBI GEO, SRA и BioProject) или результатов собственных исследований. Это обеспечивает актуальность и расширяемость БД, делая её удобным инструментом для долгосрочного использования в научных исследованиях.
Рисунок 1. Структура базы данных дифференциально экспрессирующихся генов арабидопсиса в ответ на избыточную освещенность (AraLightDEGs)
Общая информация о базе знаний и доступных в ней экспериментальных данных размещена на вкладке «About» (https://www.sysbio.ru/aralightdegs/about); вкладка «Search» (https://www.sysbio.ru/aralightdegs/search) реализует основной функционал пользовательского интерфейса поиска информации в базе данных с использованием различных фильтров по экспериментальным условиям (времени и интенсивности избыточной освещенности, возраста растений, тканей растений) и фильтрам по ДЭГ (частота идентификации, средняя экспрессия по всей выборке экспериментов) и позволяет загружать полученные пользователями результаты в табличном формате; вкладка «Download» (https://www.sysbio.ru/aralightdegs/download) позволяет пользователям осуществлять загрузку списков ДЭГ по всем экспериментальным условиям (58 таблиц), матриц экспрессии по всем экспериментам (21 таблица), а также расширенные описания информации о представленных экспериментах в табличном формате.
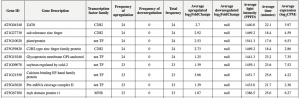
Основной формат выдачи в базе знаний AraLightDEGs на поисковой вкладке «Search» — табличный; таблица динамически генерируется во время SQL-запроса от пользователя при нажатии одной из двух кнопок быстрого поиска «Quick search for short response DEGs»/«Quick search for medium and long response DEGs» с предустановленными параметрами поиска, либо при нажатии основной кнопки поиска «Search in AraLightDEGs»; выдача табличной информации зависит от выбранных пользователем фильтров. Фрагмент таблицы выдачи из базы знаний AraLightDEGs представлен на Рисунке 2 ниже.
Рисунок 2. Фрагмент таблицы выдачи базы знаний AraLightDEGs, в которой представлены данные о ДЭГ модельного растения Arabidopsis thaliana в ответ на избыточную освещенность.
В частности, внизу поисковой страницы приводится таблица выдачи, с подразбиением на подстраницы в случае длинного вывода (1000 строк на одну подстраницу; кнопка «Subpage» позволяет их переключать). В веб-версии таблицы выдачи есть следующая информация в 11 колонках:
- Идентификаторы ДЭГ («Gene ID»), позволяющие их однозначно идентифицировать;
- Краткое функциональное описания ДЭГ («Gene Description»);
- Указание о семействе транскрипционных факторов, к которому относится ДЭГ, если относится («Transcription factor family»);
- Частота, с которой ДЭГ идентифицируется как повышающий свою экспрессию «Frequency of upregulation»;
- Частота, с которой ген идентифицируется как понижающий свою экспрессию («Frequency of downregulation»);
- Общая частота идентификации ДЭГ («Total frequency»);
- Среднее значение log2FoldChange для случаев, когда ген повышает свою экспрессию («Average upregulated log2FoldChange»);
- Среднее значение log2FoldChange для случаев, когда ген понижает свою экспрессию («Average downregulated log2FoldChange»);
- Средняя интенсивность света (микромоль * метр⁻² * секунду⁻¹), при которой наблюдались изменения экспрессии гена («Average light intensity (PPFD)»);
- Среднее время воздействия избыточной освещенности (в минутах), при котором наблюдались изменения экспрессии гена («Average time (minutes)»);
- Средний уровень экспрессии гена в логарифмической шкале (log2 counts per million, «Average expression (log2CPM)»).
Также доступна кнопка загрузки полной версии таблицы при нажатии кнопки «Download the results». Загрузка ряда колонок является опциональной и может быть включена по желанию пользователя, секция («Include in the downloadable results table»): «Gene description» — описано выше; «Transcription factor family» — описано выше; «Gene Ontology terms list» — список терминов Gene Ontology, описывающих биологические процессы, молекулярные функции и клеточные компоненты, связанные с ДЭГ; «List of experimental conditions with upregulation» — список экспериментальных условий, при которых ген повышает свою экспрессию; «List of experimental conditions with downregulation» — список экспериментальных условий, при которых ген понижает свою экспрессию; «List of log2FoldChange values of experimental conditions with upregulation» — список значений экспрессии в log2FoldChange для случаев повышения экспрессии гена в различных условиях; «List of log2FoldChange values of experimental conditions with downregulation» — список значений экспрессии в log2FoldChange для случаев понижения экспрессии гена в различных условиях.
Области возможного использования
База данных AraLightDEGs может быть использована в следующих областях:
- Генетика растений: изучение молекулярных механизмов ответа на избыточную освещенность;
- Молекулярная биология: идентификация ключевых регуляторов в генных сетях стрессового ответа растений;
- Биотехнология: разработка генетически модифицированных растений с повышенной устойчивостью к избыточной освещенности.
- Селекция растений: создание новых сортов, устойчивых к избыточной освещенности;
5. Системная биология и биоинформатика: моделирование стрессового ответа растений на транскриптомном уровне.
Степень готовности разработки к практическому применению
База данных AraLightDEGs полностью готова к практическому применению и уже используется в научно-исследовательской деятельности ИЦиГ СО РАН.
Возможный технический и (или) экономический эффект
Технический эффект использования базы данных AraLightDEGs заключается в предоставлении исследователям готовой платформы для анализа данных, что значительно ускоряет процесс изучения стрессового ответа растений. Возможный экономический эффект выражается в снижении затрат на проведение экспериментов за счет использования систематизированных данных и приоритизации генов для дальнейших исследований, что, в свою очередь, может ускорить разработки новых подходов в агробиологии и биотехнологии растений и ускоренному созданию устойчивых к световому стрессу сортов.
Сравнительные характеристики с известными разработками
База данных AraLightDEGs является разработкой, не имеющей прямых аналогов. В отличие от существующих баз данных, она предоставляет курируемую информацию о дифференциально экспрессирующихся генах Arabidopsis thaliana в ответ на избыточную освещенность, включая детальные метаданные экспериментов и удобный интерфейс для поиска и анализа.
Защита разработки
Свидетельство о регистрации №2025622410 зарегистрирована в Реестре баз данных 03.06.2025 г., Бюл. №6, дата поступления заявки: 2025621938 19.05.2025