Разработчик
ФГБНУ ФИЦ ИЦиГ СО РАН
Авторы
Пронозин А.Ю., Афонников Д.А.
Краткая характеристика
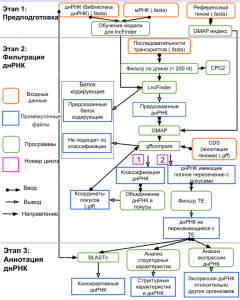
Программа предназначена для предсказания, классификации и структурно-функциональной аннотации длинных некодирующих РНК (днРНК). Программа проводит предварительную обработку данных, идентификацию днРНК, аннотацию днРНК. Входные данные программы включают сборку транскриптома, набор известных днРНК, набор известных мРНК, референсный геном исследуемого организма, аннотацию рефересного генома. Дополнительные данные: координаты мобильных элементов и экспрессия используемых библиотек в тканях. Результатами работы программы являются новые последовательности днРНК, их структурно-функциональная аннотация, анализ экспрессии в тканях, анализ тканеспецифичности, анализ гомологии и классификация на основе их выравнивания относительно белок-кодирующих генов.
Области возможного использования
Программа может быть использована для биоинформатического анализа днРНК в геномах любых организмов.
Степень готовности разработки к практическому применению
Программа готова к практическому применению.
Возможный технический и (или) экономический эффект
Улучшенная автоматизация процесса расчета, более точные подход к выявлению днРНК.
Сравнительные характеристики с известными разработками
Для идентификации днРНК в большинстве конвейеров используется программа CPC2 однако программа демонстрирует достаточно высокий уровень ошибок. В AlncRNA используется программа lncFinder, которая демонстрирует более высокую точность по сравнению с CPC2. Следующим этапом большинства конвейеров является фильтрация кандидатных последовательностей, поскольку распознавание днРНК неизбежно содержит ошибки. Большинство существующих программ применяют схожие критерии фильтрации: длина последовательности (> 200 нк.), уровень экспрессии (FPKM > 1, TPM > 1, соответственно), длина открытой рамки считывания не должна превышать 50 амк. В нашем случае мы используем систему фильтров: фильтр коротких последовательностей (потенциальные ошибки сборки и транскрипционных шум), фильтр транскриптов имеющих пересечение с мобильными элементами, фильтр транскриптов имеющих длину интрона меньше 60 нк. После выявления днРНК большинство конвейеров предлагают пользователю дополнительный анализ транскриптов: анализ консервативности между двумя исследуемыми организмами, анализ экспрессии днРНК в разных тканях, консервативности с другими организмами. Наш конвейер реализует большинство из перечисленных возможностей, а также позволяет анализировать структурные особенности днРНК (экзон-интронные структуру, особенности антисмыслового выравнивания на белок кодирующие последовательности). При этом, пользователю предоставляется свобода подачи для анализа собственных данных как по экспрессии, так и по сравниваемым внешним последовательностям днРНК.
Защита разработки
Свидетельство о регистрации № 2023665246, зарегистрирована в Реестре баз данных 13.07.2023, Бюл. № 7 , Заявка № 2023664124, дата поступления заявки: 03.07.2023.